Hay una gran variedad de herramientas bioinformáticas de Linux disponibles ampliamente utilizadas en este campo durante mucho tiempo. La bioinformática se ha caracterizado de muchas maneras; sin embargo, con frecuencia se define como una combinación de matemáticas, computación y estadística para analizar información biológica. El objetivo principal de la herramienta bioinformática es desarrollar un algoritmo eficiente para que las similitudes de secuencia se puedan medir en consecuencia.
Las mejores herramientas de bioinformática para Linux
Este artículo se ha escrito centrándose en las herramientas bioinformáticas que están disponibles en la plataforma Linux. Todas las herramientas eficientes han sido discutidas y revisadas en detalle. Además, encontrará las características esenciales, las propiedades y los enlaces de descarga de este artículo. Por lo tanto, vamos a repasarlo.
1. geWorkbench
geWorkbench se puede elaborar con el banco de trabajo del genoma es una herramienta de bioinformática basada en Java que funciona para la genómica integrada. Las arquitecturas de sus componentes facilitan complementos desarrollados específicamente que se configurarían en aplicaciones bioinformáticas complicadas. Actualmente, más de setenta complementos están disponibles para admitir, visualizar y analizar datos de secuencia.
Características de geWorkbench
- Se incluye con muchas herramientas de análisis computacional, a saber, t-test, mapas autoorganizados y agrupación jerárquica, etc.
- Se presenta con redes de interacción molecular, estructura de proteínas y datos de proteínas.
- Ofrece integración de genes y vías de anotación y recopila datos de fuentes seleccionadas para el análisis de enriquecimiento de ontologías de genes.
- En esta herramienta, los componentes se integran con la plataforma de gestión de entradas y salidas.
2. BioPerl
BioPerl es una colección de herramientas Perl ampliamente utilizada en la plataforma Linux como herramienta bioinformática para biología molecular computacional. Se utiliza continuamente en los campos de la bioinformática en un conjunto de estilo CPAN estándar. Esta herramienta de bioinformática de Linux está bien documentada y disponible gratuitamente en módulos Perl. Debido a que están orientados a objetos, estos módulos son interdependientes para realizar la tarea.
Características de BioPerl
- Desde bases de datos locales y aisladas, esta herramienta bioinformática accede a datos de secuencias de nucleótidos y péptidos.
- Manipula distintas secuencias junto con la transformación de la forma de base de datos y registro de archivo también.
- Funciona como un motor de búsqueda de bioinformática donde busca secuencias similares, genes y otras estructuras en el ADN genómico.
- Al generar y manipular alineaciones de secuencias, desarrolla anotaciones de secuencias legibles por máquina.
3. UGÉN
UGENE es un código abierto gratuito y un conjunto de herramientas bioinformáticas integradoras para Linux. Su interfaz de usuario común está integrada con las aplicaciones bioinformáticas más utilizadas y conocidas. Numerosos formatos de datos biológicos son compatibles con sus juegos de herramientas; por lo tanto, los datos se pueden recuperar de fuentes remotas. Esta herramienta bioinformática utiliza CPU y GPU multinúcleo para proporcionar el máximo rendimiento posible para optimizar sus actividades computacionales.
Características de UGENE
- Su interfaz gráfica de usuario ofrece varias funciones, por ejemplo, visualización de cromatogramas, editor de alineación múltiple y genomas visuales e interactivos.
- Allana el camino para una vista 3D en formatos PDB y MMDB junto con compatibilidad con el modo estéreo anaglifo.
- Facilita la vista de árbol filogenético, la visualización de gráficos de puntos y el diseñador de consultas puede buscar patrones de anotación complejos.
- Puede allanar el camino para un flujo de trabajo computacional personalizado para el diseñador de flujos de trabajo.
4. Biojava
Biojava es un código abierto y está diseñado exclusivamente para que el proyecto proporcione las herramientas Java necesarias para procesar datos biológicos. Funciona para una gran variedad de conjuntos de datos, por ejemplo, rutinas analíticas y estadísticas, analizadores para formatos de archivo comunes. Además, facilita la manipulación de secuencias y estructuras 3D. Esta herramienta bioinformática para Linux tiene como objetivo acelerar el desarrollo rápido de aplicaciones para conjuntos de datos biológicos.
Características de Biojava
- Incluyendo archivos de clase y objetos, es un paquete que implementa código Java para una variedad de conjuntos de datos.
- Biojava se puede utilizar en diferentes proyectos, como Dazzel, Bioclips, Bioweka y Genious, que se utilizan para diversos fines.
- Funciona para analizadores de archivos junto con los clientes DAS y el soporte del servidor.
- Se utiliza para realizar análisis de secuencias para GUI y puede acceder a bases de datos BioSQL y Ensembl.
5. Biopitón
La herramienta bioinformática Biophython desarrollada por un equipo internacional de desarrolladores y escrita en el programa python se utiliza para el cálculo biológico. Ofrece acceso en una amplia gama de formatos de archivos bioinformáticos, a saber, BLAST, Clustalw, FASTA, Genbank, y permite el acceso a servicios en línea como NCBI y Expasy.

Características de Biopython
- Se acumula con módulos de python que trabajan en hacer una secuencia con naturaleza interactiva e integrada.
- Esta herramienta bioinformática puede funcionar en diferentes secuencias, por ejemplo, traducción, transcripción y cálculos de peso.
- Esta herramienta está exclusivamente enriquecida; por lo tanto, la estructura de la proteína y el formato de la secuencia se gestionan de manera eficiente.
- Esta herramienta de bioinformática de Linux funciona para alineaciones; por lo tanto, se puede establecer un estándar para crear y tratar con matrices de sustitución.
6. InterMine
InterMine es una herramienta bioinformática de código abierto para Linux que funciona como un almacén de datos para integrar y analizar datos biológicos. Al ser un software, los usuarios pueden instalarlo en su dispositivo y hacer que los datos estén disponibles en la página web. Se cree que es una de las tablas de datos más dinámicas que puede profundizar fácilmente en los datos y facilita la forma de filtrar los datos. ¿Qué es una columna más adicional para navegar hacia la página del informe?
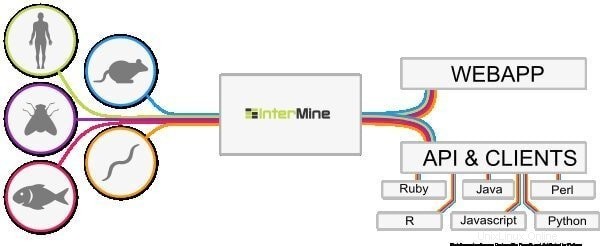
Características de InterMine
- Funciona con un único objeto, por ejemplo, un gen, una proteína o un sitio de unión, y varias listas, como una lista de genes o una lista de proteínas.
- Se puede operar en varios idiomas; por lo tanto, se pueden buscar diferentes consultas sobre información biométrica en un par de idiomas.
- En este software, hay cuatro herramientas de búsqueda disponibles:búsqueda de plantillas, búsqueda de palabras clave, generador de consultas y búsqueda de regiones.
- Admite diferentes formatos como Chado, GFF3, FASTA, GO y archivos de asociación de genes, UniProt XML, PSI XML, ortólogos In Paranoid y Ensembl.
7. IGV
IGV, elaborado como un visor de genómica interactivo, se cree que es una de las herramientas de visualización más efectivas que pueden acceder fácilmente a una base de datos de genómica extensa e interactiva. Puede ofrecer una amplia variedad de tipos de datos con anotación genómica junto con datos de secuencias de próxima generación y basados en matrices. Al igual que Google Maps, puede navegar a través de un conjunto de datos y suavizar la forma de hacer zoom y desplazarse sin problemas por todo el genoma.
Características de IGV
- Ofrece una integración flexible de amplios rangos de conjuntos de datos genómicos, incluidas lecturas de secuencias alineadas, mutaciones, números de copias, etc.
- Acelera para habilitar la exploración en tiempo real con respecto al conjunto de datos de apoyo masivo mediante el uso de formatos de archivo eficientes y de resolución múltiple.
- Entre cientos y, en cierta medida, hasta miles de muestras, permite la visualización simultánea de varios tipos de datos.
- Permite cargar conjuntos de datos de fuentes locales y remotas, incluidas fuentes de datos en la nube, para observar conjuntos de datos genómicos propios y disponibles públicamente.
8. GROMACS
GROMACS es un simulador molecular dinámico que se incluye con herramientas de análisis y construcción. Es un paquete con versatilidad y pretende trabajar en dinámica molecular; por ejemplo, puede simular la ecuación de movimiento newtoniana de cientos a miles de partículas. Fue programado para funcionar en moléculas bioquímicas en la etapa anterior, a saber, proteínas y lípidos, unidos con interacciones complicadas.
Características de GROMACS
- Esta herramienta informática de Linux es fácil de usar, contiene topologías y archivos de parámetros, y está escrita en texto no cifrado.
- No se ha utilizado lenguaje de script; por lo tanto, todos los programas funcionan con una opción de línea de comandos de interfaz simple para archivos de entrada y salida.
- Si algo sale mal, entonces se realizan muchos mensajes de error y se verifica la consistencia.
- Todos los programas se facilitan con la interfaz gráfica de usuario integrada.
9. Banco de trabajo Taverna
Taverna Workbench es una herramienta de código abierto que está programada para diseñar y ejecutar flujos de trabajo de bioinformática creados por el proyecto myGrid. Se puede integrar una variedad de software con esta herramienta, incluido el servicio web SOAP y REST. Colabora con distintas organizaciones como el Instituto Europeo de Bioinformática, el Banco de Datos de ADN de Japón, el Centro Nacional de Información Biotecnológica, SoapLab, BioMOBY y EMBOSS.
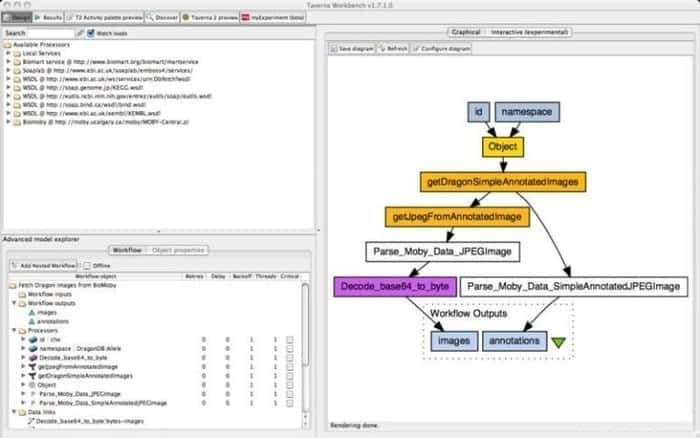
Características de Taverna Workbench
- Está completamente diseñado con el flujo de trabajo gráfico para encontrar, desarrollar y ejecutar flujos de trabajo.
- Ha sido diseñado con un flujo de trabajo completamente gráfico; además, se utilizan pestañas discretas para el diseño.
- Se proporcionan anotaciones para describir flujos de trabajo, servicios, entradas y salidas con una función de ayuda integrada.
- El flujo de trabajo utilizado anteriormente se almacena en esta herramienta, incluso si puede guardar el flujo de trabajo de entrada utilizado en el archivo.
10. RELIEVE
EMBOSS que implica European Molecular Biology Open Software Suite. Es un paquete de software que ha sido desarrollado para las necesidades de la comunidad de biología molecular. Esta herramienta de bioinformática de Linux se puede utilizar para diferentes propósitos. Por ejemplo, es funcional en varios formatos de datos automáticamente. Además, puede recopilar datos de forma secuencial desde la página web.
Características de EMBOSS
- EMBOSS se incluye con cientos de aplicaciones, a saber, alineación de secuencias y búsqueda rápida en bases de datos con patrones de secuencias.
- Además, cuenta con identificación de motivos de proteínas, incluido el análisis de dominios y el análisis de patrones de secuencias de nucleótidos.
- Su conjunto de herramientas se ha diseñado adecuadamente para abordar la aplicación y el flujo de trabajo de la bioinformática.
- Se ha programado con bibliotecas adicionales para manejar muchos otros problemas relevantes también.
11. Clúster Omega
Clustal Omega funciona con proteínas, y el ARN/ADN es un programa de alineación de secuencias múltiples diseñado para fines generales. Puede manejar de manera eficiente millones de conjuntos de datos en un tiempo razonable; además, produce MSA de alta calidad. En esta herramienta de bioinformática de Linux, existe un proceso donde el usuario requiere dejar la secuencia de archivos en el modo predeterminado. Eso se alinea y se agrupa para generar un árbol guía y, en última instancia, permite formar una secuencia de alineación progresiva.
Características de Clustal Omega
- Facilita alinear alineaciones existentes entre sí y, además, alinear una secuencia a una alineación para usar un modelo de Markov oculto.
- Hay una característica que se llama alineación de perfil externo que se refiere a una nueva secuencia de homólogos para el modelo oculto de Markov.
- Los HMM se utilizan para Clustal Omega para el motor de alineación tomado del paquete HHalign de Johannes Soeding.
- Clustal Omega permite tres tipos de entradas de secuencia:el perfil, alinear la secuencia y HMM.
12. EXPLOSIÓN
La herramienta básica de búsqueda de alineación local o BLAST se utiliza para encontrar la similitud entre las secuencias biológicas. Puede encontrar coincidencias relevantes entre secuencias de nucleótidos y proteínas y mostrar su importancia estadística. Las secuencias de consulta están estructuradas con diferentes tipos de BLAST. Lo que es más, esta herramienta se cultiva en gran medida con genes desconocidos prósperos en varios animales, y permite mapear conjuntos de datos basados en secuencias a través de análisis cualitativos.

Características de BLAST
- El nucleótido-nucleótido megaBLAST ofrece la búsqueda y optimización de tipos de secuencias muy similares.
- Además, el nucleótido-nucleótido BLASTN funciona de manera un poco diferente al buscar secuencias de distancia.
- Además, BLASTP realiza búsquedas de relación y comparación proteína-proteína, y su fórmula se utiliza para otras investigaciones diferentes.
- TBLASTN se enfoca en la consulta de nucleótidos contra el conjunto de datos de proteínas, y puede traducir la base de datos sobre la marcha.
13. Utensilios de cama
El software de bioinformática Bedtool es una navaja suiza de herramientas que se utiliza para amplios rangos de análisis genómico. La aritmética genómica utiliza esta herramienta muy ampliamente, lo que implica que puede encontrar la teoría de conjuntos con ella. Por ejemplo, las herramientas de cama facilitan el recuento, el complemento y la intersección aleatoria, la combinación de intervalos genómicos de varios archivos y la generación de un formato de genoma particular, como BAM, BED, GFF/GTF, VCF.
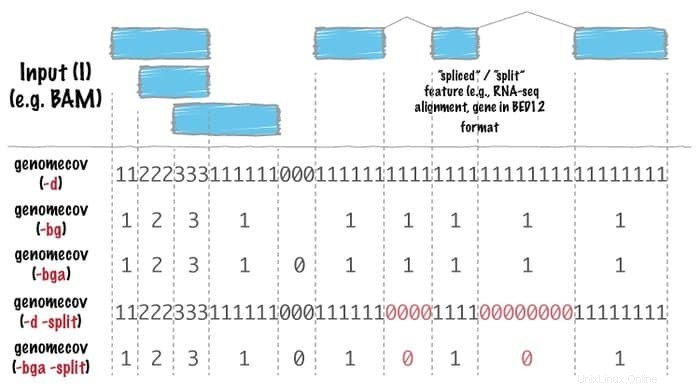
Características de Bedtools
- En esta herramienta bioinformática de Linux, cada uno está diseñado para realizar una tarea particularmente simple, por ejemplo, intersecar dos archivos de intervalo.
- El análisis complicado y sofisticado se realiza mediante el uso de una combinación de bedtools.
- Esta herramienta ha sido desarrollada en el laboratorio Quinlan de la Universidad de Utah por un grupo de investigadores.
- Dado que hay muchas opciones en esta herramienta, se puede utilizar para múltiples propósitos en el campo de la bioinformática.
14. Bioclipse
La herramienta de bioinformática Bioclipse Linux que se define con Workbench for Life Science es un software de código abierto basado en Java. Funciona en la plataforma visual que incluye quimioterapia y bioinformática Eclipse Rich Client Platform. Se presenta con una arquitectura de complemento. Eso implica la arquitectura de complementos de última generación, además, la funcionalidad y las interfaces visuales de Eclipse, como el sistema de ayuda, las actualizaciones de software también incluidas.
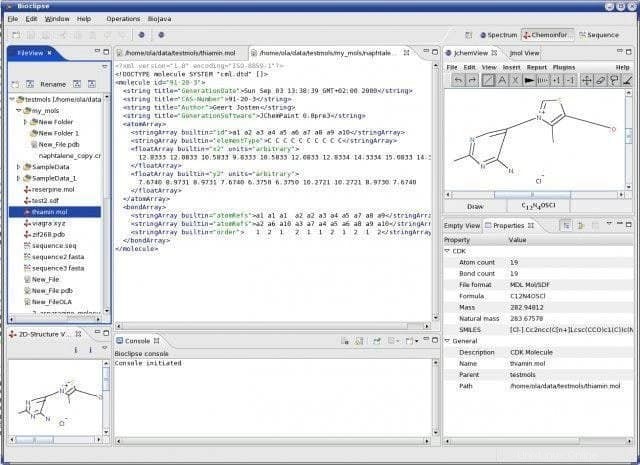
Características de Bioclipse
- Las secuencias biológicas, a saber, ARN, ADN y proteínas, se gestionan con el bioclipse.
- Biojava también ayuda a proporcionar la funcionalidad básica de bioinformática; editores gráficos para alineaciones de secuencias también.
- Se utiliza para la farmacología y el descubrimiento de fármacos junto con el sitio del descubrimiento del metabolismo.
- Finalmente, funciona en la funcionalidad de la web semántica, explora extensas colecciones de compuestos y edita estructuras químicas.
15. Bioconductor
La bioinformática utilizada ampliamente en la plataforma Linux es una herramienta bioinformática gratuita y de código abierto, utilizada coherentemente en biología médica para análisis de alto rendimiento. Utiliza principalmente programación estadística en R; sin embargo, también contiene otro lenguaje de programación. Este software está diseñado centrándose en un par de objetivos; por ejemplo, tiene como objetivo establecer un desarrollo colaborativo y garantizar el uso masivo de software innovador.
Características del bioconductor
- Este software puede analizar una variedad de datos, por ejemplo, matrices de oligonucleótidos, análisis de secuencias, citómetros de flujo y puede generar una sólida base de datos gráfica y estadística.
- Tener viñetas y documentos en cada paquete Binocular puede proporcionar una descripción textual y orientada a tareas de la funcionalidad de ese paquete.
- Puede generar datos en tiempo real sobre la micromatriz asociada y otros datos genómicos junto con metadatos biológicos.
- Además, puede analizar genes express como LIMMA, cDNA Arrays, Affy Arrays, RankProd, SAM, R/maanova, Digital Gene Expression, etc.
16. ÁNFORA
AMPHORA, que significa Aplicación de inferencia filogenómica automatizada, es una herramienta de flujo de trabajo de bioinformática de código abierto. Otra versión de AMPHORA que se llama AMPHORA2 tiene genes marcadores filogenéticos bacterianos y arqueales 104. Más importante aún, funciona para crear información entre conjuntos de datos filogenéticos y metagenéticos.
Características de AMPHORA
- Por ser un solo gen, AMPHORA2 es el más adecuado para deducir la composición taxonómica de las bacterias.
- Además, también puede inferir la composición taxonómica de las comunidades de arqueas a partir de la secuencia de escopeta metagenómica.
- Inicialmente, AMPHORA se utilizó para analizar los datos metagenómicos del Mar de los Sargazos.
- Sin embargo, hoy en día, AMPHORA2 se usa cada vez más para analizar datos metagenómicos relevantes en este sentido.
17. Anduril
Anduril es un software de bioinformática basado en componentes de código abierto para Linux que funciona para crear un marco de flujo de trabajo relacionado con el análisis de datos científicos. Esta herramienta ha sido desarrollada por el Laboratorio de Biología de Sistemas de la Universidad de Helsinki. Esta herramienta bioinformática para Linux está diseñada para permitir un análisis de datos eficiente, flexible y sistemático, especialmente en el campo de la investigación biomédica.
Características de Abduril
- Funciona en un flujo de trabajo donde se interrelacionan diferentes sistemas de procesamiento; por ejemplo; una salida de un proceso puede funcionar como una entrada de otros.
- La herramienta principal de Anduril está escrita en Java, mientras que otros componentes están escritos en diferentes aplicaciones.
- En sus diversos pasos, se desarrollan numerosas actividades, tales como; crea datos, genera informes y también importa datos.
- Su configuración de flujo de trabajo se puede realizar con un lenguaje de secuencias de comandos potente y simple, a saber, Andurilscript.
18. Servidor LabKey
LabKey Server es una opción preferida por los científicos utilizados en los laboratorios para integrar investigaciones, analizar y compartir datos biomédicos. En esta herramienta se utiliza un depósito de datos seguro que facilita las consultas, los informes y la colaboración basados en la web dentro de una amplia gama de bases de datos. Junto con la plataforma subyacente dada, se pueden agregar muchos más instrumentos científicos en esta aplicación.
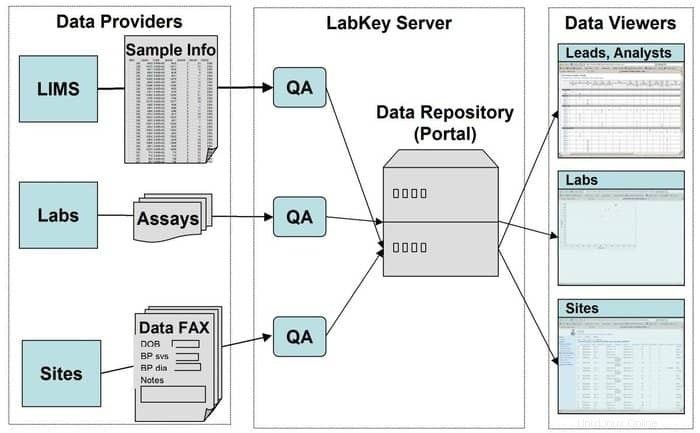
Características del servidor LabKey
- LabKey Server se presenta con todo tipo de datos biomédicos. Por ejemplo, citometría de flujo, micromatrices, espectrometría de masas, microplacas, ELISpot, ELISA, etc.
- En esta herramienta, un canal de procesamiento de datos personalizable ejecuta todas las actividades relevantes.
- Se presenta con estudios observacionales que respaldan la gestión de estudios longitudinales a gran escala de los participantes.
- La proteómica se utiliza para procesar datos de espectrometría de masas de alto rendimiento mediante una herramienta específica, a saber, X! Tándem.
19. Madre
Mothur es una herramienta bioinformática de código abierto ampliamente utilizada en el campo biomédico para el procesamiento de datos biológicos. Es un paquete de software que se usa con frecuencia para analizar el ADN de microbios no cultivados. Mothur es una herramienta bioinformática de Linux que puede procesar datos generados a partir de métodos de secuenciación de ADN, incluida la pirosecuenciación 454.
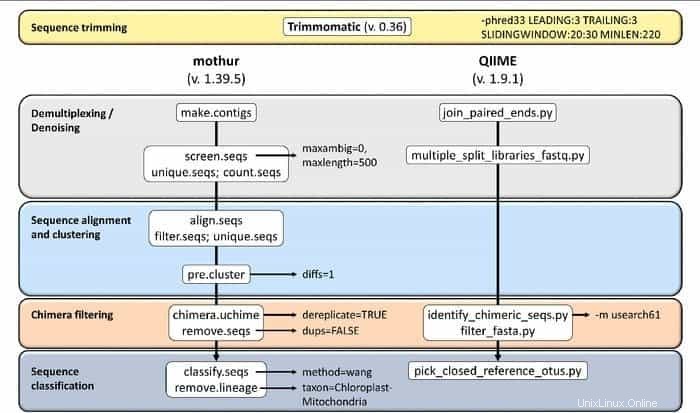
Características de Mothur
- Es un software de paquete único capaz de manejar datos de la comunidad analizando y haciendo una secuencia.
- Esta herramienta proporciona soporte de documentación comunitaria a gran escala y otra forma de soporte.
- Se cree que Mothur es la herramienta bioinformática más destacada que analiza las secuencias del gen 16S rRNA.
- Una comunidad dedicada y tutoriales están disponibles en esta herramienta para informar cómo usar Sanger, PacBio, IonTorrent, 454 e Illumina (MiSeq/HiSeq).
20. VOTCA
VOTCA significa Versatile Object-oriented Toolkit for Coarse-graining Applications, que se califica como una herramienta bioinformática eficiente con un paquete de modelado de grano grueso que analiza principalmente datos de biología molecular. Su objetivo es desarrollar técnicas sistemáticas de grano grueso junto con la simulación de carga microscópica para transportar semiconductores desordenados.
Características de VOTCA
- VOTCA se presenta principalmente con tres partes principales:el kit de herramientas de grano grueso, el kit de herramientas de transporte de carga y el kit de herramientas de transporte de excitación.
- Las tres características principales son de la biblioteca de herramientas VOTCA que implementa procedimientos compartidos.
- VOTCA utiliza métodos de grano grueso para cosechar los mejores resultados de las actividades relevantes.
- Este software se presenta con un conjunto de herramientas de transporte de excitación donde los paquetes orca DFT son compatibles en gran medida.
Reflexión final
Para resumir todo, vale la pena mencionar aquí que todas las aplicaciones bioinformáticas mencionadas anteriormente se utilizan ampliamente en este campo. Estas herramientas de bioinformática de Linux se utilizan en la ciencia médica, la farmacología, la invención de fármacos y la esfera relevante durante mucho tiempo. Finalmente, se le solicita que deje sus dos centavos con respecto a este artículo. Además, si encuentra que este artículo vale la pena, no olvide darle me gusta, compartirlo y comentarlo. Su valioso comentario será apreciado.