La biología, también conocida como ciencias de la vida, es una de las ramas fundamentales del conocimiento. Se ocupa de los procesos vitales de los organismos vivos. La historia de la investigación y el desarrollo en este campo es bastante antigua. Con el desarrollo de la tecnología informática, los hombres han creado un verdadero progreso en este campo. Desde vencer enfermedades fatales hasta resolver el misterio de un organismo vivo, la computadora es una gran compañera para los biólogos. Hay muchas herramientas biológicas de código abierto disponibles. Linux es un sistema operativo de código abierto muy personalizable que muchos investigadores prefieren. Entonces, si usted es un biólogo o un entusiasta de la biología aficionado que busca algún software de biología de Linux, es posible que desee consultar estas herramientas de biología para PC con Linux para aprovechar al máximo su estudio o investigación.
Las mejores herramientas de biología para Linux
Algunas personas tienen la idea errónea de que Linux no tiene una gran biblioteca de software. Pero se sorprenderá de que en la categoría de software basado en la educación y la investigación, Linux sigue siendo imbatible. Es porque la mayoría de los científicos e investigadores están con el movimiento del software de código abierto.
Por lo tanto, obtiene una amplia colección de herramientas de biología para Linux. Son gratuitos y no menos que cualquier software pago. Aquí he hecho una lista curada de 15 herramientas de diferentes tipos para que no tengas que molestarte en encontrarlas. Si lee este artículo completo, espero que encuentre el mejor software para el sistema Linux, que se adapte a sus necesidades.
1. RELIEVE
La explicación del nombre del software es European Molecular Biology Open Software Suite. Es una herramienta de biología de código abierto para Linux hecha para personas interesadas en el campo de la biología. EMBOSS es una poderosa herramienta de análisis secuencial. Es algo así como un paquete completo de herramientas que las características y posibilidades de esta herramienta están más allá de toda explicación.
Características clave de EMBOSS
- Puede rastrear y recuperar rápidamente datos secuenciales de la web.
- EMBOSS se utiliza para la alineación de secuencias, la identificación de motivos de proteínas, el análisis de patrones de secuencias de nucleótidos, etc.
- Tiene una biblioteca integrada para lanzar nuevas herramientas de código abierto.
- Se incorpora una herramienta de presentación avanzada para la publicación rápida de los datos recuperados.
- Puede manejar cadenas, comparar patrones, procesar listas e indexar bases de datos usando bibliotecas de programación adicionales.
- La función de integración es útil para sincronizar con otras herramientas populares.
2. NAMD
NAMD es un programa de simulación desarrollado especialmente para simular grandes sistemas biomoleculares. Esta herramienta de biología para Linux es tan poderosa que puede procesar millones de átomos a la vez en forma paralela. Charm++ es un lenguaje basado en C++ que se usa para escribir este programa. NAMD utiliza un entorno de tiempo de ejecución llamado Converse para ejecutarse en sistemas paralelos basados en clústeres, lo que ayuda a procesar grandes cantidades de datos biológicos a la vez.

Características clave de NAMD
- La simulación de la estructura molecular se prepara utilizando Visual Molecular Dynamics.
- Admite diferentes tipos de archivos de entrada, incluidos X-PLOR, CHARMM, AMBER, etc.
- NAMD utiliza la integración de varios pasos de tiempo para el análisis numérico.
- Los usuarios pueden seleccionar entre una amplia gama de opciones de simulación dinámica.
- Es compatible con el procesamiento acelerado por GPU.
- Esta herramienta es compatible con el muestreo general basado en réplicas a través del módulo de variables colectivas.
3. GROMACS
GROMACS no es solo una herramienta más de simulación biológica; más bien, es un paquete de software completo con herramientas integradas de construcción y análisis. Esta versátil herramienta de biología para Linux puede realizar análisis y simulaciones de miles a millones de partículas biológicas. Fue desarrollado principalmente para el análisis de productos químicos biológicos como proteínas y lípidos. Pero ahora, también se usa en campos de investigación no biológicos.
Características clave de GROMACS
- Esta herramienta es dos o tres veces más rápida que sus competidores.
- El código del software está altamente optimizado para un procesamiento de datos más rápido.
- Gromacs es bastante fácil de usar. Los códigos de error están escritos con texto sin formato para facilitar su comprensión.
- El extenso manual de usuario de esta herramienta está disponible de forma gratuita en formato de papel electrónico.
- Puede almacenar datos de trayectoria en un método compacto.
- Tiene algunas herramientas integradas para el análisis de trayectoria. Los usuarios no necesitan escribir ningún código para este propósito.
- Cuenta con un generador de topología totalmente automatizado para proteínas, que es muy útil.
4. DMV
VMD es un programa avanzado de visualización biomolecular desarrollado para Linux. Un programa de visualización molecular es principalmente un programa para mostrar datos moleculares con gráficos 3D. VMD puede leer y analizar archivos PDB o Protein Data Bank y representarlos de forma gráfica estructurada. Incluso puede simular moléculas para diferentes condiciones y casos. Por lo tanto, se ha convertido en un programa muy útil para los investigadores profundos de la biología.
Características clave de VMD
- Puede utilizar la potencia de GPU externa de la computadora.
- El desarrollador no ha aplicado ninguna limitación para el número de moléculas u otros parámetros. ¡La memoria RAM es tu límite!
- Los usuarios pueden generar fácilmente archivos PDF a partir de la salida 3D estándar con la herramienta integrada.
- VMD puede utilizar el sistema de visualización estéreo siempre que lo tenga.
- La amplia biblioteca de lectores de archivos integrados admite hasta 60 formatos de archivo diferentes.
- Los investigadores pueden escribir sus comandos de rutina utilizando el lenguaje Tcl.
5. simuPOP
SimuPOP no es otra herramienta de biología ordinaria para Linux. Más bien es un entorno de simulación de genética de poblaciones en el tiempo futuro. Puede analizar y simular cualquier problema relacionado con la población. De ahí que los investigadores en el campo de la biología utilicen esta herramienta para simular la propagación de enfermedades complejas. simuPOP utiliza Python como lenguaje de secuencias de comandos central.
Características clave de simuPOP
- Tiene la opción de adjuntar campos de información a los individuos de una población.
- Tiene límites numéricos para el número de conjuntos homólogos de cromosomas u otros parámetros.
- Cuenta con más de 70 operadores integrados para análisis de población.
- La interfaz de secuencias de comandos avanzada ofrece a los usuarios la posibilidad de personalizar este programa.
- simuPOP tiene un completo sistema de documentación para principiantes.
6. MÚSCULO
MUSCLE es la abreviatura del nombre original del software MUSCLE MU ltiple S secuencia C comparación por L og-E expectativa Es una herramienta de biología muy popular para Linux, que se utiliza para crear múltiples alineaciones de secuencias de aminoácidos o nucleótidos. Además, su mejor precisión y mejor velocidad lo mantienen por delante de otros competidores como ClustalW2 o T-Coffee. Se considera uno de los programas más rápidos en esta categoría.

Características clave de MUSCLE
- Es compatible con tres funciones diferentes de puntuación del perfil de proteínas.
- MUSCLE proporciona funciones de optimización de anclaje y diagonal.
- El popular formato basado en texto FASTA se utiliza en esta herramienta como archivos de entrada y salida.
- Cuenta con un beneficio adicional que puede generar archivos de salida en diferentes formatos populares como LUSTALW, MSF, HTML, etc.
7. Vistas al mar
SeaView es un software normal de alineación de secuencias múltiples. Pero su especialidad es que tiene una interfaz gráfica de usuario muy buena y fácil de usar. Este paquete se usa como backend para otras herramientas populares como Clustal Omega, Gblocks y PhyML. Fast Light Toolkit, comúnmente conocido como FLTK, potencia la interfaz de usuario de este programa.

Características clave de SeaView
- Es compatible con la mayoría de los formatos de archivo para la secuenciación de proteínas y ADN, incluidos NEXUS, MSF, CLUSTAL, FASTA, PHYLIP, etc.
- Los usuarios pueden importar archivos de formato FASTA externos para algoritmos de alineación.
- Puede dibujar árboles filogenéticos y generarlos en diferentes formatos comunes como PDF, SVG, EPS, etc., para imprimir o publicar.
- SeaView tiene un descargador integrado para descargar secuencias genéticas de Internet.
8. ÁRBOL-ROMPECABEZAS
TREE-PUZZLE es el nuevo nombre del software PUZZLE. Es una herramienta de biología muy popular para Linux. Originalmente es un algoritmo de búsqueda de árbol basado en consola que se utiliza para el análisis de grandes conjuntos de datos. Este paquete de software TREE-PUZZLE puede reconstruir árboles utilizando los algoritmos descritos por Strimmer y von Haeseler.
Características clave de TREE-PUZZLE
- Utiliza algoritmos desconcertantes de cuarteto.
- Esta herramienta puede asignar estimaciones de soporte para cada sucursal interna automáticamente.
- TREE-PUZZLE puede construir árboles ingresando conjuntos de árboles dados por el usuario.
- Tiene algunas herramientas para realizar pruebas estadísticas en los conjuntos de datos.
- Puede estimar parámetros y distancias por pares.
9. Vista de árbol X
Es una herramienta biológica de código abierto para construir árboles filogenéticos. El software de construcción de árboles es muy importante en el campo de la biología. Es por eso que se considera una buena herramienta de biología de Linux. Puede leer archivos de árbol con diferentes formatos de archivo.
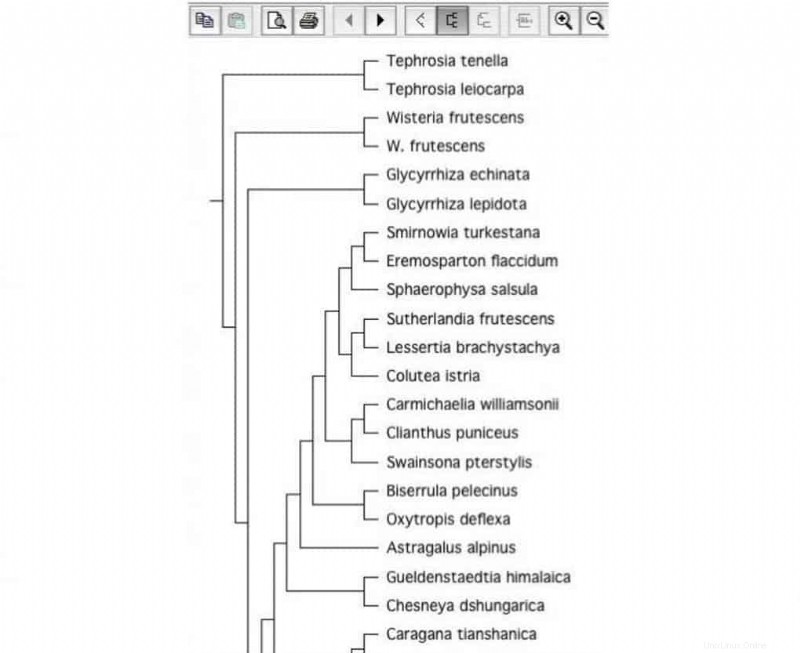
Características clave de TreeView X
- Tiene una rica GUI basada en la biblioteca wxWidgets C++.
- Puede exportar árboles en diferentes formatos de archivo basados en imágenes.
- TreeView X tiene una opción de impresión avanzada incorporada que ayuda a formatear los números de papel de impresión de acuerdo con las necesidades del usuario.
- La función de arrastrar y soltar aumenta la productividad al usar esta herramienta.
10. UGÉN
Es un software de biología de código abierto para Linux. UGENE se utiliza para el análisis de varios datos biológicos. Hoy en día, se utiliza principalmente para la secuenciación del genoma. Los datos analizados se pueden almacenar en el almacenamiento de la computadora o incluso en una base de datos de laboratorio compartida. La interfaz gráfica de usuario de esta herramienta ayuda a los usuarios a operar sin ningún conocimiento previo de codificación. Aparte de la GUI, también tiene una interfaz de línea de comandos heredada para trabajar.
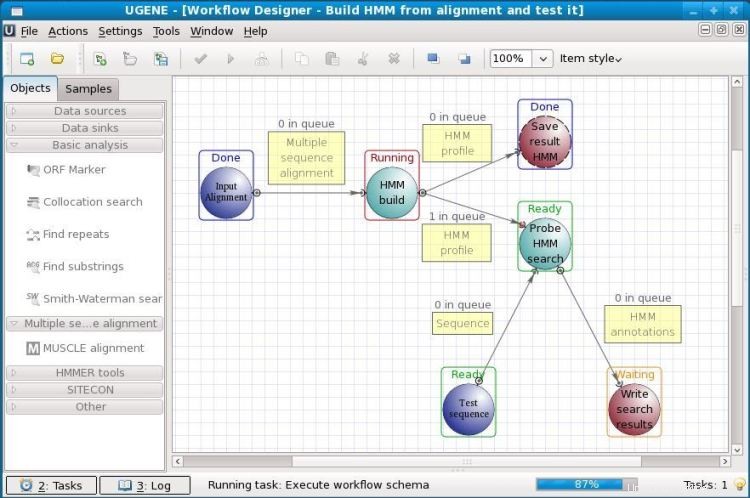
Características clave de UGENE
- Los usuarios pueden crear y anotar secuencias de proteínas fácilmente.
- Puede utilizar los múltiples núcleos de la CPU host y puede utilizar una tarjeta gráfica discreta.
- Tiene integración incorporada con servidores bioinformáticos populares como PDB, NCBI, etc.
- UGENE tiene una herramienta integrada Primer3 para el diseño de un cebador de PCR.
- Cuenta con un visor de cromatogramas avanzado.
- Esta herramienta puede buscar señales complejas con ExpertDiscovery.
11. Primer3
Primer3 es uno de los software de biología más populares para Linux. Es una herramienta de biología gratuita y de código abierto para Linux bajo licencia GNU. Esta herramienta se utiliza para seleccionar el cebador de una secuencia de ADN. Esta herramienta también tiene una interfaz de usuario web alternativa llamada Primer3 Plus para aquellos que no desean instalarla localmente.
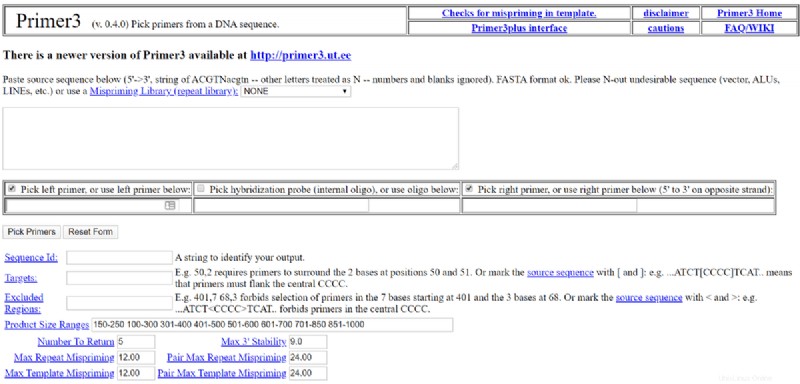
Características clave de Primer3
- Los usuarios pueden importar/cargar archivos de secuencia en casi cualquier formato de archivo popular.
- Las secuencias se pueden pegar en texto sin formato.
- Tiene muchas funciones de personalización en la categoría de configuración general y avanzada.
- Los usuarios pueden ingresar la calidad de la secuencia en esta herramienta.
- Hay una pestaña dedicada a los pesos de penalización en esta herramienta.
12. Navegador de genoma integrado
Como sugiere el nombre, es un navegador de genoma para su escritorio. Es una herramienta de biología gratuita y de código abierto. Este software de biología para Linux puede buscar secuencias genómicas en Internet. Por supuesto, puede buscar estos datos bioinformáticos particulares a través de su navegador habitual. Pero créame, este navegador dedicado hará que su flujo de trabajo sea mucho más rápido. Esta herramienta se basa en Genoviz SDK, una biblioteca de Java.
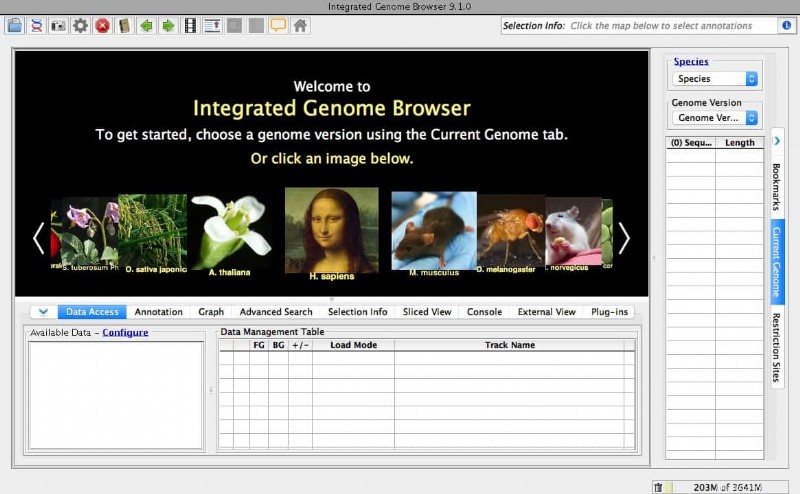
Características clave del navegador del genoma integrado
- Esta herramienta puede leer datos de muchos formatos de archivo, incluidos BAM, BED, Affymetrix CHP, FASTA, GTF, PSL, etc.
- Los usuarios pueden exportar la salida a cualquier formato imprimible como SVG, PNG o incluso PDF fácil de usar.
- Funciones de zoom y desplazamiento dinámicas y en tiempo real.
- Admite servicios web de estilo REST para funciones de anotación.
13. LÁMPARAS
LAMMPS es una de las herramientas biológicas de código abierto más populares. The abbreviation stands for “L arge-scale A tomic/M olecular M assively P arallel S imulator.” It is a general-purpose molecular dynamics software. But nowadays, it is highly used in the field of biological research. It is developed and maintained by Sandia National Laboratories. This Linux biology software uses Message Passing Interface or MPI protocol for parallel communication among researchers.
Key Features of LAMMPS
- It uses an efficient data structure named the Verlet List for keeping track of nearby particles.
- It can utilize the full potential of a parallel computing system by dividing the simulation domain into smaller subdomains and distributing them for each processor.
- This tool is highly portable because it is made in C++.
- Built-in support for CUDA and OpenCL GPU rendering system.
- Users can easily extend new features and functions.
14. Madre
Mothur is a well-known Linux biology software among scholars. This software project was initiated by Dr. Patrick Schloss et al. Many publications of biological research have cited this software so far. This open-source tool is a very efficient bioinformatics data processor. It is mostly used for the DNA analysis of uncultured microbes.
Key Features of Mothur
- It can process data generated from several DNA sequencing methods.
- Almost all the popular methods are supported by this tool, including 454 pyrosequencing, Illumina HiSeq and MiSeq, Sanger, PacBio, and IonTorrent.
- No other tools can beat Mothur in analyzing 16S rRNA gene sequences.
- It is maintained regularly by a group of well-known scholars of biology.
15. PathVisio
PathVisio is a free and open-source biology tool for Linux. It is used for drawing, editing, and analyzing biological pathways. It has many useful features built-in with the package. Users can also install additional features via plugins. This tool is based upon Java, and this is why it can easily be installed on any platform, including Linux.
Key Features of PathVisio
- Advanced drawing and annotation tools for pathways.
- It can even analyze different types of biological pathways.
- PathVisio has built-in integration with WikiPathways for easier publishing.
- The open-source tool Cytoscape can be easily integrated with this tool.
- It can be integrated with other programming languages via PathVisioRPC.
Reflexiones finales
As you can see, there are numerous tools for the different purposes needed in the field of biology. Biology is a vast field of knowledge and research. So it is obvious that you won’t need to use all of the tools mentioned above. If you try out this curated list of Linux biology software, you will get to know which will suit your works best. And, if you have any favorite software in this category, you can let others know by commenting below.